Нечай Н.Л., Оспанкулова
Г.Х., Тоймбаева Д.Б., Нестеров В.А.
ТОО
«Научно-исследовательский институт переработки сельскохозяйственной продукции»,
Казахстан
E-mail:natasha_nechai@mail.ru
Изучение видового состава
возбудителей молочнокислого брожения кумыса
В последние годы наблюдается повышенный интерес
к потреблению традиционных кисломолочных продуктов, для получения которых используют
широкий спектр различных сырьевых материалов, микроорганизмов и технологий.
В настоящее время в мире насчитывается более чем
3500 различных ферментированных пищевых продуктов и напитков. Большинство из
этих продуктов производятся в Азии, Африке и в странах Ближнего Востока [1-4]. Производство
кисломолочных напитков в домашних условиях осуществляется на естественных
заквасках, в промышленных условиях - на чистых культурах [5].
Кумыс – казахский национальный кисломолочный
напиток, получаемый из свежего кобыльего молока. Кобылье молоко имеет свои
сугубо специфические особенности - это альбуминовое молоко, с большим содержанием
молочного сахара, малыми размерами жировых шариков, значительным содержанием
витамина С. В среднем кобылье молоко содержит 2,1% белка (из которых 1,2%
казеина и 0,9% сывороточного белка), 1,2% жира, 6,4% лактозы, 0,4% золы [7,8]. В
связи с этим кумыс обладает высокими питательными и биологическими свойствам [6]. В результате соблюдения технологии приготовления
кумыса в продукте накапливается молочная кислота (0,7-1,8%), этанол (0,6-2,5%)
и диоксид углерода (0,5-0,9%) [9]. Углекислый газ придает пенистый вид продукту
и специфический аромат.
В
зависимости от стадии брожения получают продукты трех категорий называемые
"слабый", "средний" и "крепкий" кумыс. Мягкий
кумыс содержит молочной кислоты - 0,6-0,8% и спирта - 0,7- 1,0%, средний -
0,8-1,0% молочной кислоты и 1,1-1,8% спирта, сильный - имеет 1,0-1,2% молочной кислоты и 1,8-2,5%
спирта.
В настоящее время в исследованиях, описывающих
процесс брожения при приготовлении кумыса, отмечаются несколько основных группы
микроорганизмов. Эти лактобациллы: такие как Lb. delbrueckii подвид bulgaricus, Lb. salivarus, Lb. buchneri, Lb. рlantarum, Lb. casei, Lb. helveticus, Lb. fermentum; дрожжи такие как Saccharomyces lactis, Torula, Kluyveromyces lactis, Saccharomyces unisporus [12,13].
В лаборатории биопереработки зерновых культур
ТОО «КазНИИПСХП» были проведены исследования по изучению состава микробиоты
кумыса.
Молочнокислые бактерии и дрожжи были выделены из
образцов кумыса домашнего приготовления
с использованием естественных заквасок. Отбор проб кумыса проводили на
территории Акмолинской области
(Аршалынский район с. Семеновка; с. Коргалжын,
Аккольский район с. Кенес, г.
Ерейментау).
Пробы кумыса, отобранные для анализа, имели
серовато – белый цвет, кислый вкус, привкус углекислого газа. Что
соответствовало качественным показателям (таблица 1).
С целью выделения возбудителей молочнокислого
брожения в лабораторных условиях был проведен посев проб кумыса на
агаризованную питательную среду МРС – 1 методом серийных разведений, инкубировали
при температуре 30°C. Учет численности микроорганизмов проводили через 72 ч
культивирования. Количество микроорганизмов в кумысе варьировало от 11 до 155
тыс/мл (рисунок 1) в зависимости от происхождения кумыса, качества кобыльего молока,
длительности сбраживания, условия хранения.
В зависимости от времени созревания, пробы кумыса,
произведенных в с. Аршалы и с. Кенес, были «слабой» и «средней» категорий,
согласно ОСТ 1461148-83 «Кумыс натуральный». Вероятно, с этим и связаны
различия в численности микроорганизмов.
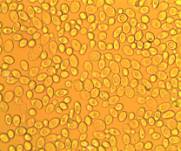
При микроскопировании чистых культур было
установлено что в кумысе присутствовали бактерии и дрожжи различных родов (рисунок
2). Дрожжи имели грушевидную форму клеток, часто с
перетяжками, находящиеся в стадии почкования (рисунок 2 В). Колонии
дрожжеподобных грибов на среде МРС достигали диаметра 5-9 мм, на 7
сутки культивирования имели молочно-белую или бежевую окраску, ровную, бархатистую
поверхность колоний.

Рисунок 1 – Общая
численность микроорганизмов в пробах кумыса, тыс/мл
1-
Аршалынский район с. Семеновка, 2- г. Ерейментау, 3- п. Коргалжын, 4-
Аккольский район с. Кенес


А Б В
Рисунок
2 – Бактерии (А,Б) и дрожжи (В), выделенные из проб кумыса, х1000
Среди исследователей нет единого мнения
относительно роли отдельных видов дрожжей в кумысе. До сих пор остается спорным
вопрос о значении в спиртовом брожении дрожжей, не сбраживающих лактозу. Вместе
с тем, в литературе имеются указания, что эти виды дрожжей, хорошо развиваются
в молоке за счет сбраживания глюкозы и галактозы, образующихся в начальной
стадии молочнокислого брожения [5].
Чистые культуры бактерий, выделанные из кумыса
подвергли молекулярно – генетической идентификации. Идентификация культур
рабочей коллекции была осуществлена методом определения прямой нуклеотидной
последовательности фрагмента 16S rRNA
гена, с последующим определением
нуклеотидной идентичности с последовательностями депонированными в
международной базе данных Gene Bank, а также построением филогенетических
деревьев с нуклеотидными последовательностями референтных штаммов.
ДНК исследуемых бактерий выделяли методом Kate
Wilson [10]. Концентрацию
ДНК измеряли спектрофотометрическим
методом с использованием спектрофотометра NanoDrop, при длине волны 260 нм, а
также проводили качественную оценку ДНК электрофоретическим методом.
Реакция
ПЦР была выполнена с универсальными
праймерами [11] 8f 5’ – AgAgTTTgATCCTggCTCAg-3 и 806R- 5’ ggACTACCAgggTATCTAAT. ПЦР
программа была выполнена с применением
амплификатора GeneAmp PCR System
9700 (Applied Biosystems).
Очистку ПЦР продуктов от не связавшихся
праймеров проводили, ферментативным методом
используя, Exonuclease I (Fermentas) и щелочную фосфатазу (Shrimp
Alkaline Phosphatase, Fermentas) [12].
Реакцию секвенирование проводили с применением
BigDye® Terminator v3.1 Cycle Sequencing Kit (Applide Biosystems) согласно инструкции производителя, с последующим
разделением фрагментов на автоматическом генетическом анализаторе 3730xl DNA
Analyzer (Applide Biosystems).
Нуклеотидные последовательности 16S rRNA гена
идентифицируемых штаммов были анализированы и объединены в общую
последовательность в программном обеспечении SeqScape 2.6.0 (Applide Biosystems). После чего были удалены
концевые фрагменты (нуклеотидные последовательности праймеров, фрагменты,
имеющие низкий показатель качества), что
позволило нам получить нуклеотидную последовательность протяженностью более 650 п.н., которые были идентифицированы в GeneBank по алгоритму BLAST. Дополнительно,
было проведено построение филогенетического древа с нуклеотидными последовательностями 16S rRNA гена референтных
штаммов данных видов (рисунок 3).
Построение филогенетических древ проводили с
использованием программного обеспечением Mega 3.1 [13], выравнивание
нуклеотидных последовательностей проводили используя алгоритм ClustalW,
построение филогенетических деревьев проводили с использованием метода ближайших соседей (Neiighbor-Joining
NJ).
В результате генетической
идентификации 1 группа бактерий была идентифицирована как L. paracasei; 2 группа - L. kefiri; 3 группа - L. farciminis; 4 группа - L. diolivorans, 5 группа - L. brevis.
Результаты исследований показали, что количество
микроорганизмов в кумысе зависит от времени созревания продукта. Количество, видовой состав микроорганизмов и соотношение в кумысе бактерий и дрожжей
зависит от стадии
брожения. На ранних стадиях созревания кумыса отмечается значительное
количество бактерий, более поздние стадии
созревания кумыса характеризуются значительным количеством дрожжей.
В кумысе произведенном в Акмолинской области
Республики Казахстан среди молочнокислых бактерий преобладают виды Lactobacillus paracasei, Lactobacillus kefiri, Lactobacillus farciminis, Lactobacillus diolivorans, Lactobacillus brevis.
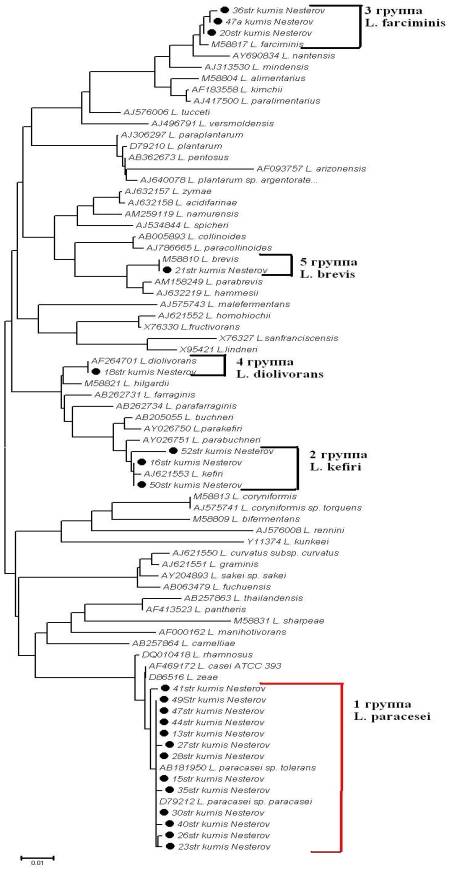
Рисунок 3. Филогенетическое дерево
построенное на основании анализа нуклеотидной последовательности 16S rRNA гена.
Литература:
1. Simango, C.
(1997). Potential use of traditional fermented foods for weaning in Zimbabwe.
//Soc. Sci. Med. 44: 1065–1068.
2. Gadaga, T.H.,
Mutukumira, A.N., Narvhus, J.A., and Feresu, S.B. (1999). A review of
traditional fermented foods and beverages of Zimbabwe.// Int. J. Food Microbiol.
53: 1–11.
3. Blandino, A.,
Al-Aseeri, M.E., Pandiella, S.S., Cantero, D., and Webb, C. (2003).
Cereal-based fermented foods and beverages. Food Res. Int. 36: 527– 543.
4. Chang J.-H.,
Shim Y.Y. Cha S.-K., Chee K.M. (2010) Probiotic characteristics of lactic acid
bacteria isolated from kimchi. // Journal of Applied Microbiology 109 (2010)
220–230]
5. Гашева, Марзият Асланчериевна Разработка
технологии напитка смешанного брожения на основе традиционных кисломолочных
продуктов// Диссертация кандидат технических наук Ставрополь 2010
162 c
6. Marconi, E.,
and Panfili, G. (1998). Chemical composition and nutritional properties of
commercial products of mare milk powder. J. Food Compos.
Anal.11: 178–187.
7. Коханов М.А. (2009) Научно-практическое
обоснование способов повышения эффективности производства продукции коневодства
при табунном содержании лошадей// Диссертация д.с.-х.н.,- Волгоград, - 2009,
334с.
8. Дамдинсурэнгийн Наранцэцэг (2000) Влияние пищевых структурообразователей на
биохимические изменения в белковом комплексе кумыса при хранении// Диссертация
к.т.н. Краснодар - 2000 , -207 c
9. Tamime, A.Y.,
Muir, D.D., and Wszolek, M., (1999). Kefir, koumiss and kishk. Dairy Ind. Int.,
64: 32–33.
10. Wilson K. Preparation of
genomic DNA from bacteria. Current
Protocols in Molecular Biology. Editors (Editors Ausubel, F. M., Brent, R.,
Kingston, R. E., Moore, D. D., Seidman, J. G., Smith, J. A., et al.). - New York: Wiley, 1987.- 650 p.
11. Vegas E. Z.
S., Nieves B., Araque M., Velasco E.,
Ruiz J., Vila J. Outbreak of Infection
With Acinetobacter Strain RUH 1139 in an Intensive Care Unit // Infection
control and hospital epidemiology. – 2006. - Vol. 27, № 4. - P. 397 – 404.
12. Werle E.,
Schneider C., Renner M., Völker M., Fiehn W. Convenient single-step, one
tube purification of PCR products for direct sequencing // Nucleic Acids Res. –
1994. – Vol. 22. - P. 4354-4355.
13. Kumar S.,
Tamura K., Nei M. MEGA3: Integrated
software for Molecular Evolutionary Genetics Analysis and sequence alignment //
Briefings in bioinformatics. – 2004. – Vol. 5, №2. – P. 150–163.